Mikrobiologie | Analyseverfahren
SCHNELLER ANTIBIOTIKARESISTENZEN BESTIMMEN
Seite 1/1 2 Minuten
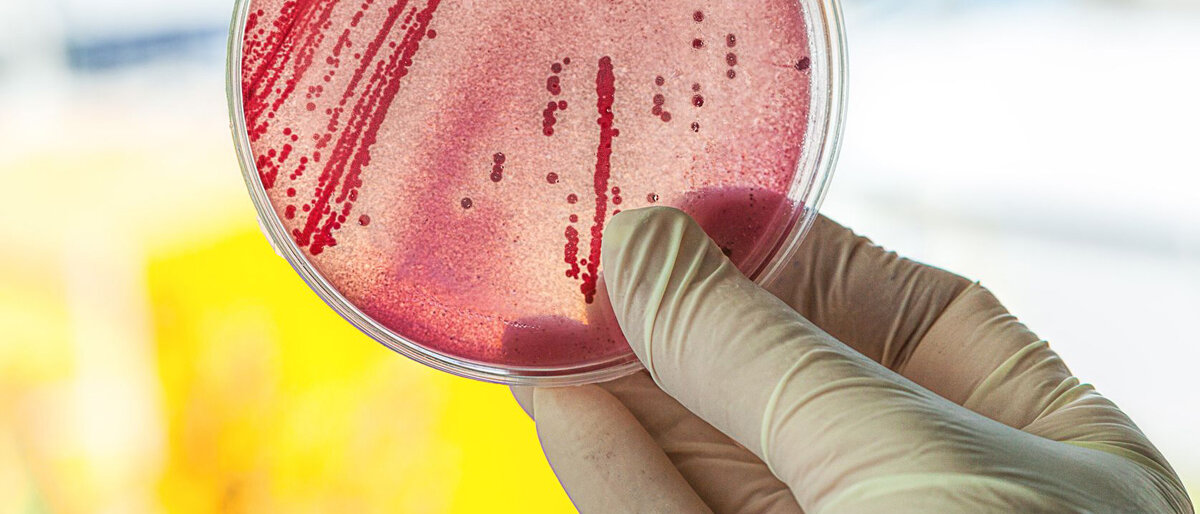
Egal ob Harnwegs-, Atemwegs- oder Wundinfektion – sind Bakterien beteiligt, ist ein schnelles Eingreifen notwendig, um Komplikationen zu verhindern. Das Beste wären natürlich passgenau auf die Mikrobe zugeschnittene Antibiotika, doch bis zum Erregernachweis dauert es mindestens einen Tag, in manchen Fällen kann dies zu lang sein und man haut lieber mit der „Breitband-Keule“ drauf. In der Regel folgt aktuell nach einer Probenentnahme (zum Beispiel Abstrich, Blutentnahme), das Anlegen einer Kultur in einem geeigneten Nährmedium. Bei optimalen Wachstumsbedingungen (bestimmte Temperatur, Nahrung, Zeit) vermehren sich die Bakterien, sodass anschließend verschiedene bakterizide und bakteriostatische Arzneistoffe an ihnen auf ihre Wirksamkeit hin untersucht werden können (Antibiogramm). Ein neu entwickeltes mikrobiologisches Testverfahren soll diesen Weg verkürzen, indem es die Erreger auf Resistenzmerkmale untersucht.
Die neue Methode des Forscherteams des Instituts für Medizinische Mikrobiologie der Universität Münster basiert auf der MALDI-TOF-Massenspektrometrie, mit der es bereits möglich ist, Erreger zu identifizieren.
Bei der Analysemethode genügen geringe Mengen einer Bakterienkultur. Diese werden in einer bestimmten Matrix gelöst und auf einen Träger aufgebracht. Durch den Laserbeschuss im Massenspektrometer wird die Probe ionisiert und die in der Probe enthaltenen Proteine können nach ihrem Ladungs-Masse-Verhältnis aufgetrennt werden. Das daraus resultierende Muster (Massenspektrogramm) kann mit bereits bekannten Daten verglichen werden und dient so der Identifizierung der Erreger. Es gleicht also einem Fingerabdruck des Bakteriums. Der Fortschritt, den die Wissenschaftler nun erreicht haben, besteht darin, anschließende langwierige Sensibilitätstest (welches Antibiotikum kann eingesetzt werden, worauf reagiert der Keim sensibel) zu umgehen und somit die Analysezeit deutlich zu verkürzen. Dazu entwickelten sie basierend auf dieser Methodik eine universelle Schnellmethode zur Empfindlichkeitsklärung. Damit können gleichzeitig mehrere Antibiotika an dem Keim getestet werden – eine enorme Zeitersparnis.
Zurzeit arbeitet das wissenschaftliche Team eng mit dem Bremer Medizintechnik-Unternehmen Bruker Daltonik zusammen, um das technische Verfahren weiter zu verfeinern und schnell zur Marktreife zu bringen. Durch die Einführung dieser Innovation zur Identifikation von Resistenzmerkmalen könnten Ärzte bald schneller die optimale Antibiotikatherapie auswählen und im Falle multiresistenter Keime geeignete Hygiene- und Eradikationsmaßnahmen veranlassen. Ein daraus resultierender geringerer Einsatz von Breitbandantibiotika könnte zudem die Zunahme der Entwicklung resistenter Erreger-Stämme reduzieren.
Farina Haase,
Apothekerin, Volontärin
Quelle: Informationsdienst Wissenschaft